


Siguiente: Predicción de estructura secundaria de proteínas
Subir: Propiedades moleculares que se pueden calcular
Anterior: Estabilidad de un dúplex de ADN
Índice de Materias
La reacción en cadena de la polimerasa
tiene muchas aplicaciones en biología, medicina y biotecnología. Todas estas aplicaciones dependen del
empleo de parejas de cebadores o primers, oligonucleótidos que sirven de puntos de arranque de la actividad
polimerasa. En concreto, la extensión de los cebadores se da en su extremo 3'. Por estas razones es deseable
que los cebadores usados en una PCR cumplan ciertas condiciones, todas ellas calculables desde su
secuencia:
- deben tener longitudes de entre 19 y 25 nucleótidos
- deben ser ricos en contenido en GC
- el contenido en GC de los cebadores de la misma reacción debe ser similar
- deben ser complementarios a las regiones deseadas, aunque admiten degeneraciones
- las temperaturas de fusión de los primers usados en la misma reacción deben ser similares, normalmente
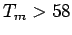
- es requisito que las secuencias de los cebadores no formen horquillas (hairpins) internas
ni sean complementarias entre si.
En la Web hay ejercicios para aprender a diseñar primers como
éste
y éste otro.
El artículo de Rose et al. (2003), disponible aquí, presenta una
de las aplicaciones del diseño de primers degenerados.
La tarea de hoy incluye:
- usar el servidor web de CODEHOPS con este
alineamiento,
- para ello primero hay que cortar el alineamiento en bloques conservados y
- luego ejecutar codehops
- finalmente, es necesario emparejar codehops que podríamos usar en una PCR, que evaluaremos con este
programa python checkprimers.py



Siguiente: Predicción de estructura secundaria de proteínas
Subir: Propiedades moleculares que se pueden calcular
Anterior: Estabilidad de un dúplex de ADN
Índice de Materias
Centro de Ciencias Genómicas/UNAM, México 2006-7